5 Dentro e fuori R
v1.2.1 3/11/2023
5.1 Cosa c’è da imparare in questo capitolo.
Aprire, salvare e condividere file di dati è una necessità costante per chi si occupa di statistica e analisi dei dati. Le tabelle di dati possono essere salvate in una varietà di formati, alcuni gestibili da tutti i software (file di testo, a spaziatura fissa o delimitati, come i file .dat, .txt, .tab, .tsv, .csv)103 e altri proprietari (.xlsx, .numbers, etc., ma anche fogli di calcolo on line come Google Fogli). Nella tua vita precedente ti sei abituat* a usare le funzioni e i menu del sistema operativo del tuo computer o del tuo programma preferito per fogli di calcolo per navigare nel file system, creare, aprire e chiudere cartelle, cercare, aprire e salvare file. Per quanto tu possa sfruttare molte delle funzioni di RStudio per fare praticamente le stesse cose (vedi dopo), se hai deciso di fare lo sforzo di leggere questo libro è per fare qualcosa di più e, soprattutto per poterlo fare in maniera riproducibile e, se possibile, automatizzata.
Come al solito, sei liber* di scegliere il tuo percorso. Se non hai tempo, o voglia, leggi solo le prime tre sezioni (5.2; 5.3; 5.4) e lascia il resto per dopo. Nei capitoli successivi troverai diversi esempi di comandi per leggere e salvare file di testo.
Se sei interessat* ad operazioni più complesse, leggi anche le Appendici 5.8 e 5.9.
5.2 Ogni cosa al suo posto: working directory, progetti, etc.
Quando inizi una sessione di R (aprendo direttamente R o aprendo RStudio), il software sceglie automaticamente una cartella di lavoro (working directory), che diventerà il luogo in cui verranno cercati o salvati i file, a meno che tu non fornisca indicazioni diverse. Di default la tua working directory è la tua home come utente104. Per conoscere quale è la tua working directory scrivi nella console105:
>getwd()Nel mio caso (uso un progetto, vedi dopo), il risultato è:
>"/Users/eugenio/pigR"Quindi, la cartella su cui sto lavorando è pigR, che è contenuta nella mia home, eugenio, che a sua volta è contenuta nella cartella Users del mio file system. Io uso MacOS, quindi il delimitatore è /, mentre nei sistemi Windows è \.
Puoi cambiare la working directory con il comando setwd().
Guarda cosa succede usando questi comandi: copiali e incollali in uno script vuoto o scrivili nella console (ho già omesso il prompt) ed eseguili per vedere l’effetto che fa:
# stampo a console la wd e attribuisco il nome lamiawd all'oggetto restituito
# dalla funzione getwd()
lamiawd <- getwd()
# che tipo di oggetto è?
# cambio la wd. Il comando non è specifico per il mio sistema, perché .. indica la
# cartella che contiene la cartella su cui sto lavorando
setwd("..")
getwd()
# riporto al wd a quella originale
setwd(lamiawd)Ovviamente, puoi anche fare le stesse operazioni usando i menu di R studio (figura 5.1), che ti consentono di scegliere la working directory usando dei semplici menu interattivi.
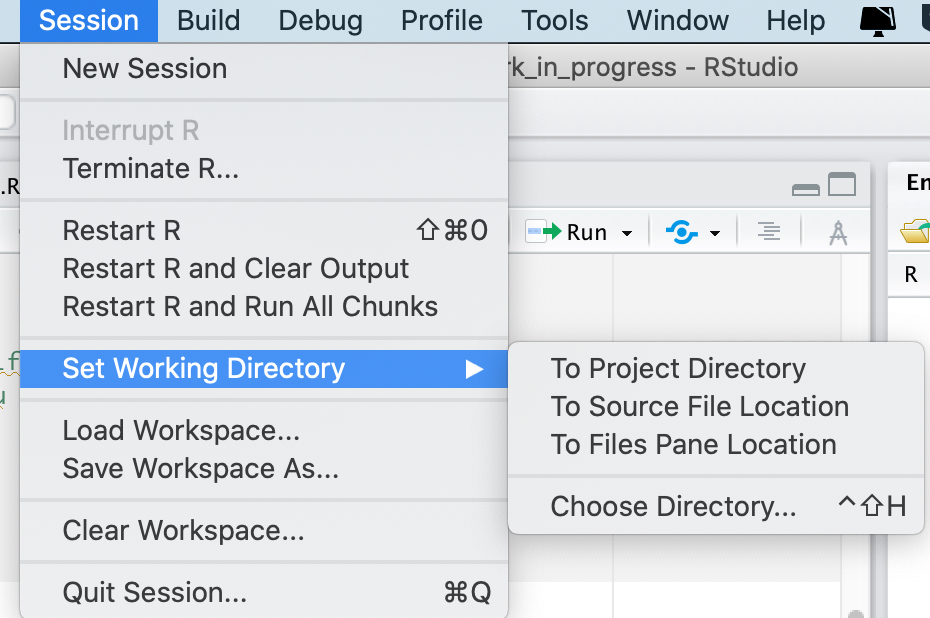
Figura 5.1: Scegliere la working directory da menu.
Mentre quando si lavora in modo interattivo è abbastanza facile fare queste operazioni, quando si lavora con uno script, un documento interattivo o un rapporto R Markdown (vedi capitoli successivi) questo non è possibile. Inoltre, diventa laboriosissimo indicare con chiarezza dove si è lavorato o riprodurre il flusso di analisi o, peggio ancora, farlo riprodurre a qualcun altro.
Fortunatamente, RStudio mette a disposizione un sistema molto più efficiente: la creazione di progetti. Un progetto è fondamentalmente una cartella in cui è contenuto tutto il materiale (script, file di dati, file di immagini, storia, etc.) per un determinato progetto di lavoro. RStudio si occupa di creare tutta una serie di cartelle e file nascosti che, quando riapriamo un progetto, ci permette di riprendere esattamente dal punto dove avevamo lasciato (specialmente se ci ricordiamo di salvare l’ambiente di lavoro quando usciamo da R). Basta trasferire l’intera cartella a qualcun altro perché all’apertura del progetto tutto ritorni esattamente nello stato in cui lo hai lasciato tu. Geniale!!!!
Ci sono 3 modi diversi di creare un progetto:
usando il menu in alto a destra in RStudio (vedi figura 5.2)
usando l’icona di accesso rapido in alto a sinistra (un piccolo cubo con la lettera R e un
+bianco in campo verde)usando il menu
File -> New project
Il progetto può essere creato in una cartella (directory) esistente o in una nuova cartella106.
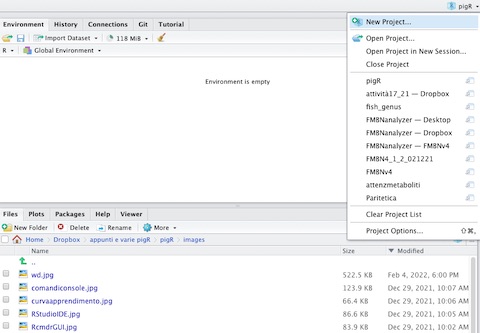
Figura 5.2: Il menu progetti.
Una volta creato un progetto, il suo nome comparirà sotto la voce File -> Open Recent Projects e nel menu progetti (figura 5.2). E’ anche possibile aprire un progetto semplicemente con un doppio click.
5.3 Facciamola semplice: il menu Import Dataset.
Come ho già detto da qualche parte, se sei fortunat* lavorerai prevalentemente con tabelle di dati, create da te con un programma di foglio di calcolo e potrai salvarle sia in formato testo (sai farlo, vero?) che nel formato proprietario della app che stai usando (o in formato MS Excel, magari creato usando Fogli di Google, o Numbers, o chissà che). In questo caso, dovresti sapere come è fatto il tuo file e non dovresti avere troppi problemi a importarlo, perché sicuramente avrai seguito i miei consigli sulla struttura delle tabelle (4.1.3) e sui nomi delle variabili (4.3). In altri casi potresti trovarti ad aprire file di cui non conosci la struttura, non sai se contengono o meno righe di commento, come sono indicati i missing data, etc. Fortunatamente per te, RStudio mette a disposizione nel pannello dell’ambiente un po’ di menu semplici e diretti per aprire file in formato .RData o in formato testo o Excel.
Proviamo subito con un semplice esercizio. Per sicurezza esporterai prima un dataset di R in diversi formati (è semplice, basta seguire le istruzioni) e poi lo importerai in maniera interattiva.
Crea un progetto in una cartella esistente o in una nuova cartella107
Crea un nuovo script con
File->New->R ScriptCopia e incolla questo codice (dovresti già aver installato i pacchetti necessari nei capitoli precedenti, altrimenti segui le istruzioni presentate nel Paragrafo 2.2.10)
library(tidyverse)
library (readxl)
library(vcd)
library(openxlsx)
data("Arthritis")
# salva un file delimitato da , con . come separatore decimale
write_csv(Arthritis, file = "Arthritis_comma.csv")
# salva un file delimitato da ; con . come separatore decimale
write_csv2(Arthritis, file = "Arthritis_semicolon.csv")
# salva un file delimitato da tabulazioni con . come separatore decimale
write_tsv(Arthritis, file = "Arthritis_tab.txt")
# salva un file .xlsx
write.xlsx(Arthritis, file = "Arthritis.xlsx")Ora fai girare lo script seguendo uno dei metodi descritti nel Capitolo 2. Come risultato, i file che hai salvato dovrebbero apparire nella tua working directory. Useremo questi file per provare a importare i dati con la funzionalità ‘Import Dataset’ di RStudio. E’ possibile anche aprire lo stesso menu da File->Import Dataset.
Ora proviamo a importare uno qualsiasi dei file. Nell’esempio successivo userò Arthritis_semicolon.csv, ma tu puoi sceglierne un altro, ovviamente scegliendo di conseguenza le opzioni migliori.
Nelle Figure 5.3 e 5.4 sono illustrati i passaggi da seguire.
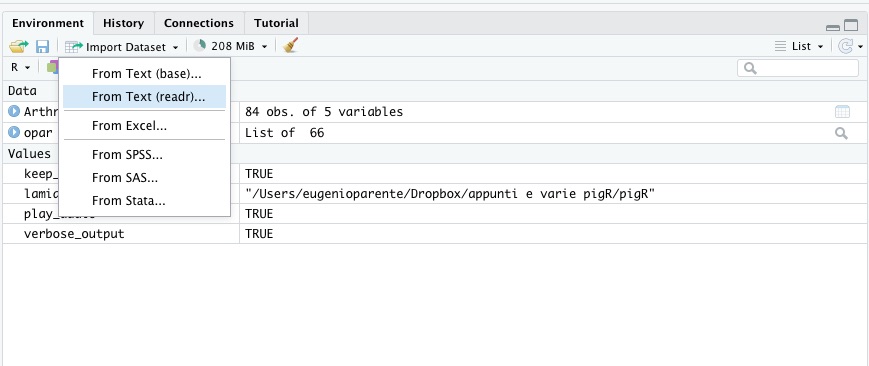
Figura 5.3: Aprire il menu Import Dataset.
Prima di tutto seleziona Import Dataset -> From Text (readr). readr è uno dei pacchetti dei tidyverse ed è ottimizzato per aprire e salvare in modo veloce ed efficiente file di testo.
La finestra che otterrai è mostrata nella figura 5.4. puoi scegliere il file che vuoi aprire navigando fra le cartelle con il tasto
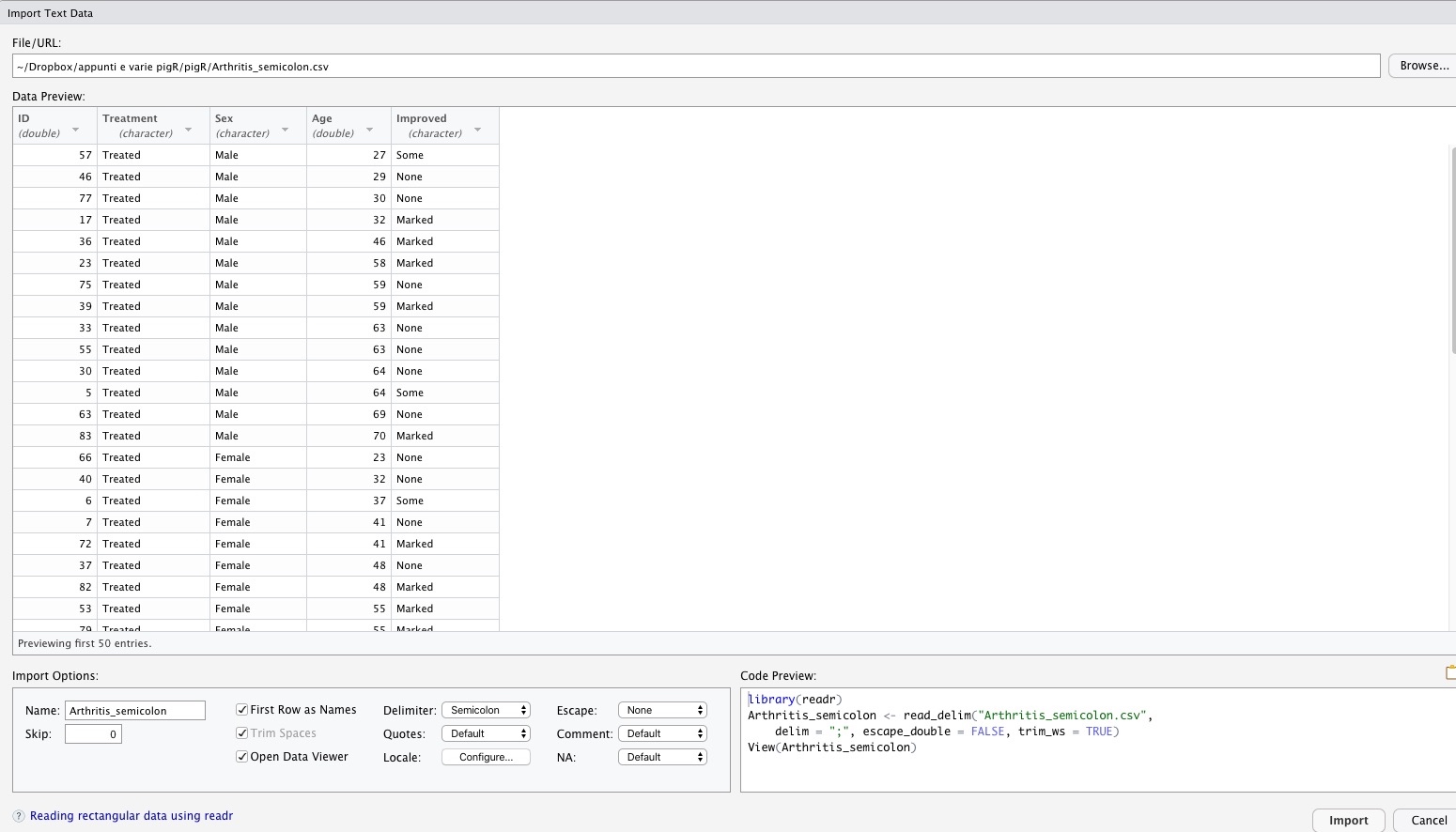
Figura 5.4: Importare file di testo.
Le funzioni dei campi, checkbox e menu dovrebbero essere abbastanza intuitive. Ad ogni modo:
Nameconsente di assegnare un nome all’oggetto importatoSkipconsente di selezionare il numero di righe da scartare all’inizio del file: alcuni file di testo contengono commenti seguiti poi dai datiFirstRow as Namesindica, se selezionato, che la prima riga contiene i nomi delle variabili (di solito è così; in alternativa R userà dei nomi di default)Trim spacesse selezionato eliminerà eventuali spazi vuoti prima o dopo il contenuto delle celleOpen data viewer, se selezionato, aprirà la tabella nelViewerdopo l’importazioneDelimiterconsente di scegliere il delimitatore delle colonne: se non siete certi provate e vedrete come il contenuto si distribuisce in colonne con le diverse opzioniQuotespermette di determinare come vengono trattate le virgolette contenute nei campiLocaleconsente di selezionare le opzioni che determineranno lingua, sistemi di data, delimitatore decimale etc.109Escapeconsente di scegliere il carattere di escape, che serve per importare correttamente caratteri speciali, come/Commentconsente di scegliere il carattere che precede i commentiNAconsente di scegliere il modo in cui sono codificati, nel file di importazione, i missing data; anche qui, potete fare un po’ di prove e vedere che succede, magari usando la barra di scorrimento verticale per dare un’occhiata.
I comandi corrispondenti a queste azioni sono generati in automatico e possono essere copiati ed incollati in uno script (per rendere riproducibile l’azione).
library(readr)
Arthritis_semicolon <- read_delim("Arthritis_semicolon.csv",
delim = ";", escape_double = FALSE, trim_ws = TRUE)
View(Arthritis_semicolon)Se non lo si indica specificamente con un’opzione110, readr determina automaticamente il tipo delle colonne dalle prime 1.000 osservazioni.
In realtà le opzioni di readr sono molto più numerose e forniscono un’enorme flessibilità nell’importazione dei dati. Le funzioni di readr sono in genere superiori alle corrispondenti funzioni di R base (vedi qui il perché). Tuttavia, è importante ricordare che le funzioni di readr creano o usano tibbles e le tibbles non hanno, fra le altre cose, nomi di riga. Sono quindi inadatte ad alcune strutture di dati, come le matrici e gli array o i data frame con nomi di riga, che possono essere utili in diverse situazioni.
Lo stesso menu Import Dataset permette di importare molti altri tipi di file e di usare, se preferite, le funzioni di base. Sentitevi liberi di sperimentare, magari con i file che avete appena creato.
5.4 Gestire file di testo con readr.
Lavorare in maniera interattiva con il menu Import Dataset va bene finché non devi automatizzare il tuo flusso di lavoro o condividerlo con altri. In questo caso, devi abituarti a usare i comandi generati da questo menu in maniera programmatica111. Rispetto alle funzioni di base è veloce ed efficiente e può leggere direttamente file da posizioni sul web. Prova a scrivere nella console:
study_metadata <- read_tsv("https://tinyurl.com/34u6a9md")Il comando importa la tabella presente all’indirizzo indicato e assegna l’oggetto112 al nome study_metadata. E’ addirittura possibile decomprimere e aprire file compressi.
readr offre una serie di comandi per importare file di testo delimitati in tibble e per salvare tibble come file di testo (tabella 5.1)
| Delimitatore | per importare | per esportare | Commenti |
|---|---|---|---|
| qualsiasi | read_delim() | write_delim() | richiedono più configurazione |
| tabulazione | read_tsv() | write_tsv() | legge sia .tsv che .txt |
| virgola | read_csv() | write_csv() | è quello più comune |
| punto e virgola | read_cvs2() | write_csv2() |
Tieni presente che la , è usata nei paesi che usano come delimitatore decimale il punto e il ; è usato nei paesi che usano come delimitatore decimale la virgola.
Questi sono i comandi più comuni. I comandi di importazione hanno la struttura:
il_mio_oggetto <- read_delim(file, ...)dove ... sono le opzioni (leggi l’aiuto delle funzioni!). Come hai visto file può essere un percorso di file113 o anche una URL. Il risultato è una tibble114. Aprire file da una URL può essere particolarmente utile se lavori con una versione cloud di R].
I comandi per l’esportazione hanno la struttura:
write_delim(il_mio_oggetto, file, ...)Fra le altre cose, offrono l’opzione append che, con append = T, permette di aggiungere dati alla fine di un file invece di sovrascriverlo.
readr può importare e esportare file molto grandi (e mostrare una barra di avanzamento mentre lo fa), ma gli oggetti importati non possono eccedere il limite di RAM utilizzabile (vedi dopo per qualche alternativa).
Infine, readr può importare file delimitati da spazi (read_table) o con campi di larghezza fissa (read_fwf) e molte altre cose. Vedi le pagine di aiuto o vai al sito di readr per saperne di più.
5.5 Tutti i comandi (o quasi) per importare file di testo…
Esistono molti modi di pelare un gatto e con R, l’importazione e l’esportazione di file di testo possono essere ottenute con diversi pacchetti. La tabella successiva ti offre un piccolo confronto.
| Delimitatore | base/ultils | readr | data.table |
|---|---|---|---|
| qualsiasi | read.table() | read_delim() | fread() |
| write.table() | write_delim() | ||
| tabulazione | read.delim() | read_tsv() | |
| write.delim() | write_tsv() | | | |
| virgola | read.csv() | read_csv() | |
| write.csv() | write_csv() | ||
| punto e virgola | read.cvs2() | read_csv2() | |
| write.csv2() | write_csv2() |
L’uso del . invece di _ non è la sola differenza dei comandi base e quelli di readr.
I comandi base:
possono solo leggere da file
creano data frame (e quindi conservano i nomi di riga)
possono salvare sia matrici che data frame
hanno dei default un pochino meno intuitivi
Per esempio, read.csv2() assume, contemporaneamente, che il separatore sia il punto e virgola e che il delimitatore decimale sia il la virgola. Leggi l’aiuto dei singoli comandi per saperne di più.
data.table::fread() è una funzione molto potente e veloce per l’importazione di file di testo da file o da URL. Esistono molti confronti fra questa funzione e quelle di R e base e readr: prova a cercare da sol* con Google.
Come ultima cosa: in R è possibile aprire “connessioni” con file o URL per leggere o scrivere dati con vari sistemi. Questo argomento è un po’ oltre gli obiettivi di questo corso; consulta la Sezione 5.10 per saperne di più.
5.6 Altri formati di dati.
Se collabori con altri analisti e ricercatori ti potrebbe capitare di dover importare ed esportare in altri formati. A parte formati specialistici (come i formati utilizzati per i network, gml, o quelli usati per gli alberi filogenetici), che sono gestiti da pacchetti specifici, è abbastanza frequente importare ed esportare dati in un formato leggibile da Microsoft Excel115 o formati specifici per altri software statistici (SPSS, SAS, Stata, Systat).
A parte questo, R ha un’incredibile quantità di pacchetti e funzioni per importare dati da database in diversi formati e per ottenere dati da social media e dal web. Vedi nella Sezione 5.10 per qualche suggerimento specifico.
5.6.1 Da e per Excel.
Il modo forse più efficiente e coerente con il tidyverse per importare tabelle da Excel è usare le funzioni read_excel() (giudica il formato dall’estensione), read_xls() o read_xlsx() (leggono i file del formato corrispondente) di readxl. Quest’ultimo è un pacchetto del tidyverse che non viene caricato esplicitamente con library(tidyverse) e va quindi installato e caricato a parte. Se hai salvato Arthritis in formato .xlsx prova a eseguire questi comandi116:
>arthritis_xlsx <- read_excel("Arthritis.xlsx")
>arthritis_xlsxCome vedi, i comandi del tidyverse hanno bisogno di pochissima configurazione e producono delle tibble.
Curiosamente, readxl non ha funzioni per esportare i dati in Excel: readr offre due funzioni, write_excel_csv() e write_excel_csv2() che permettono di produrre file .csv più compatibili con Excel (che in realtà può importare facilmente file di testo, ma è sicuramente preferibile farlo da menu).
Il pacchetto openxlsx offre funzioni che permettono, in maniera più articolata di importare117 ed esportare file in formati .xls e .xlsx. Prevedibilmente, i nomi di questi funzioni sono read.xlsx() e write.xlsx(). Le opzioni di questi comandi sono piuttosto articolati. Prova a leggerne l’aiuto. Se non ricordi come, vai al Capitolo 3.
5.6.2 Altri formati: foreign e Hmisc e haven.
I pacchetti foreign, Hmisc e haven forniscono vari comandi per importare file di dati da altri software di analisi statistica. La Tabella 5.3 mostra alcuni esempi.
| Software | foreign | Hmisc | haven |
|---|---|---|---|
| STATA | read.dta() | read_dta() | |
| SPSS | read.spss() | spss.get() | read_sav() |
| Systat | read.systat() | ||
| SAS | read.ssd() | sas.get() | read_sas() |
| MINITAB | read.mtp() |
Tutti questi comandi restituiscono data frame. foreign fornisce solo alcuni comandi per l’esportazione. Fai riferimento all’aiuto per la sintassi e le opzioni. Le funzioni di Hmisc hanno in genere default più semplici. haven è un pacchetto del tidyverse e quindi restituisce tibbles.
5.6.3 Un richiamo: file .Rdata e .rds
R ha i propri formati per il salvataggio di file di dati in formati binari, estremamente compatti. Ne ho parlato in precedenza (Capitolo 4). Ecco un piccolo riassunto con esempi:
save.image(file = "ilmiofile.Rdata"): salva un’immagine dell’ambiente di lavoro, con gli oggetti e i loro nomisave(Arthirtis, Arthritis_semicolon, file = "imieioggetti.Rdata"): salva una serie di oggetti (indicati con i loro nomi o come stringhe di caratteri118); gli oggetti vengono salvati con i loro nomiload(file = "ilmiofile.Rdata"): carica nell’ambiente di lavoro tutti gli oggetti contenuti nel file Rdata. copompresi i loro nomisaveRDS(Arthirtis, file = "Arthritis.rds"): salva un singolo oggetto. L’oggetto salvato può essere ricaricato attribuendogli un altro nomereadRDS(file = "Arthritis.rds"): legge un singolo oggetto, che di solito viene attribuito a un nome, per esempioarthritis_2 <- readRDS(file = "Arthritis.rds")
Nota che readr fornisce due versioni alternative a readRDS() e saveRDS(), rispettivamente read_rds() e write_rds()119.
5.6.4 E’ andato tutto bene?
Ma come si fa a sapere se l’importazione è andata bene? Se tutte le variabili sono state importate nel formato (numerico, testo, logico, etc.) corretto? Se dati mancanti, commenti o righe vuote iniziali, finali etc. hanno causato problemi? Se ci sono spazi vuoti prima e dopo il testo di una colonna? Se i caratteri speciali sono stati improtati correttamente?
Innanzitutto, se non conosci con certezza la struttura della tabella che vuoi importare (magari semplicemente perché la ha creata qualcun altro) è sempre una buona idea dare una sbirciatina usando un programma di lettura di semplici file di testo (come Textedit in MacOS)
Per tabelle molto piccole (<1000 righe, qualche decina di colonne) è abbastanza facile aprire la tabella nel Viewer120 dopo l’importazione.
Per tabelle più grandi ci sono varie strategie (e stampare la tabella in console non è una di quelle):
usare il comando
pillar::glimpse()121, che fornisce una rappreentazione compatta mostrando i tipi delle variabili e alcune osservazioni;usare il comando
summary(), che fornisce alcune statistiche riassuntive sulle colonne (adattandole a seconda del tipo dei dati nella colonna)fare qualche elaborazione grafica (vedi Capitolo 6)
Prova tu stess* scrivendo nella console i seguenti comandi e confrontandone l’output (ricorda che Arthritis è un data frame):
>View(Arthritis)
>Arthritis
>class(Arthritis)
>Arthritis_2 <- as_tibble(Arthritis)
>Arthritis_2
>glimpse(Arthritis_2)
>summary(Arthritis)5.7 Lavorare con i nomi dei file e le directory.
Capire l’uso dei percorsi per file e cartelle in R non è facile se sei abituat* a gestire questi compiti nelle interfacce grafiche del tuo sistema operativo. RStudio ti offre diverse opzioni per usare interfacce semplici e immediate (vedi sopra e vedi Capitolo 2).
Anche se imparare a usare i percorsi dei file non è necessario se vuoi lavorare in modo interattivo, è essenziale sia per ottenere analisi riproducibili e generalizzabili (riutilizzabile con altri tipi di file dello stesso tipo) e per lavorare con i report di RMarkdown122. Nell’esempio successivo troverai un certo numero di comandi utili a gestire file e cartelle in maniera programmatica. Prova a usare questi comandi sul tuo sistema e nota le differenze nell’output. Se sei arrivat* fino a qui non sei del tutto pigr* e quindi dovresti fare, prima o poi, uno sforzo per leggere l’aiuto di questi comandi per sfruttarli al meglio.
Prima di tutto, i percorsi di file e directory sono specifici per ogni piattaforma; prova questo:
## [1] "/Users/eugenio/Library/CloudStorage/Dropbox/appunti e varie pigR/pigR_bookdown"Come sono separati i nomi delle cartelle nel percorso? Con / (caratteristico dei sistemi Unix) o \ (caratteristico dei sistemi Windows e usato come carattere escape in R)?
L’uso di delimitatori diversi per i percorsi pone un problema: quando apri e salvi i file devi definire i percorsi. Ammettiamo che tu voglia preparare un esempio con una cartella contenente uno script e alcuni file: se tu definissi i percorsi, anche relativi (alla cartella di lavoro, vedi sopra), sul tuo sistema, lo script non funzionerebbe necessariamente in un altro sistema operativo. Un modo per definire i percorsi indipendente dalla piattaforma è file.path(). In questo comando gli elementi del percorso (a loro volta singole cartelle o gruppi di cartelle) sono separati da “,”. Prova ad eseguire il comando descritto nell’esempio successivo sul tuo sistema e osserva cosa succede.
## [1] "/Users/eugenio/Library/CloudStorage/Dropbox/appunti e varie pigR/pigR_bookdown/DataL3"E’ particolarmente utile ottenere una lista dei file e cartelle in una determinata cartella. Il comando relativo è list.files(). Guarda gli esempi successivi e prova ad eseguire gli stessi comandi sul tuo sistema.
## chr [1:23] "_book" "_bookdown_files" "_bookdown.yml" "_main_files" ...## [1] "_book"
## [2] "_bookdown_files"
## [3] "_bookdown.yml"
## [4] "_main_files"
## [5] "_main.Rmd"
## [6] "01--Introduzione.Rmd"
## [7] "02--Interfacce-e-ambienti.Rmd"
## [8] "03--Chiedere-aiuto.Rmd"
## [9] "04--Dati_oggetti_funzioni_linguaggio.Rmd"
## [10] "05--Dentro_e_fuori.Rmd"
## [11] "06--Grafica.Rmd"
## [12] "07--Descrivere-con-i-numeri.Rmd"
## [13] "08--Creare_report.Rmd"
## [14] "09--Lottare_con_i_dati.Rmd"
## [15] "10--Credits.Rmd"
## [16] "images"
## [17] "index.Rmd"
## [18] "lavori"
## [19] "pigR_bookdown.Rproj"
## [20] "pigR_epub_book"
## [21] "pigR_gitbook"
## [22] "pigR_reviewer"
## [23] "renderepub.R"# ora uso . per accedere gerarchicamente i livelli del file system
# nella cartella di lavoro
list.files(".")## [1] "_book"
## [2] "_bookdown_files"
## [3] "_bookdown.yml"
## [4] "_main_files"
## [5] "_main.Rmd"
## [6] "01--Introduzione.Rmd"
## [7] "02--Interfacce-e-ambienti.Rmd"
## [8] "03--Chiedere-aiuto.Rmd"
## [9] "04--Dati_oggetti_funzioni_linguaggio.Rmd"
## [10] "05--Dentro_e_fuori.Rmd"
## [11] "06--Grafica.Rmd"
## [12] "07--Descrivere-con-i-numeri.Rmd"
## [13] "08--Creare_report.Rmd"
## [14] "09--Lottare_con_i_dati.Rmd"
## [15] "10--Credits.Rmd"
## [16] "images"
## [17] "index.Rmd"
## [18] "lavori"
## [19] "pigR_bookdown.Rproj"
## [20] "pigR_epub_book"
## [21] "pigR_gitbook"
## [22] "pigR_reviewer"
## [23] "renderepub.R"## [1] "bookdown-demo-main"
## [2] "BUP-Scheda_Autore_pigR.docx"
## [3] "BUP-Scheda_Autore.docx"
## [4] "capitolo_2_le_GUI.docx"
## [5] "Capitolo_3_aiutoR.docx"
## [6] "capitolo_4_elementi_base_linguaggio.docx"
## [7] "da_spostare_in_pigR_bookdown"
## [8] "Errori battitura.docx"
## [9] "immagini_dim_originali"
## [10] "Indice pigR.docx"
## [11] "notabene.rtf"
## [12] "pexels-mark-stebnicki-2252541 small.jpeg"
## [13] "pexels-mark-stebnicki-2252541_300.jpg"
## [14] "pexels-mark-stebnicki-2252541.jpg"
## [15] "pigR_bookdown"
## [16] "pigR_note.docx"
## [17] "pigR_Rmd"
## [18] "Proposta di pubblicazione, tempi rapidi.rtf"
## [19] "PROPOSTA EDITORIALE HOEPLI.pdf"
## [20] "Re_ proposta editoriale per BUP.rtf"
## [21] "screenshotsRstudio"Prova ad eseguire il comando successivo dalla console:
> list.files(".", recursive = T)Cosa succede? Prova ad attribuire il risultato di questo comando ad un nome. Che oggetto è? Che usi potresti farne?
Determinare con esattezza il percorso di un file può essere difficile, specialmente se il file si trova “annidato” molto profondamente nel tuo sistema. A parte le funzionalità del pannello dell’ambiente aprire e salvare file e quelle del pannello File di RStudio per esplorare il tuo file system, un comando interessante è file.choose(), che apre un menu interattivo per la scelta di un file e ne restituisce il percorso. Prova ad eseguire questi comandi nella tua console:
> il_nome_del_file <- file.choose()Due comandi interessanti per ottenere, dato un percorso, la directory che contiene il file di interesse e il nome del file sono:
> dirname(il_nome_del_file)
> basename(il_nome_del_file)Guarda che succede ora. Prova a ricostruire quello che fanno questi comandi usando l’aiuto:
## [1] "_book" "_bookdown_files" "_bookdown.yml"
## [4] "_main_files" "_main.Rmd" "01--Introduzione.Rmd"la_mia_wd <- getwd()
il_nome_della_mia_wd <- basename(la_mia_wd)
# la tilde abbrevia il percorso a una cartella, path.expand() lo espande
file.path("~","il_nome_della_mia_wd")## [1] "~/il_nome_della_mia_wd"## [1] "/Users/eugenio/il_nome_della_mia_wd"## character(0)R fornisce alcuni utili comandi per verificare l’esistenza di cartelle in una determinata posizione o per crearle. Prova a leggere l’aiuto:
>?dir.createProva ad eseguire questo breve esempio: il codice cerca una cartella dal nome “lavori” all’interno della cartella di lavoro e, se non c’è, la crea:
la_mia_wd <- getwd()
if(!dir.exists(file.path(la_mia_wd,"lavori"))) dir.create(file.path(la_mia_wd,"lavori"))5.8 Appendice 1. Un piccolo dettaglio sulla sintassi.
Nei capitoli precedenti e in questo hai visto moltissimi comandi/funzioni, ciascuno dei quali può avere da poche a molte opzioni, alcune delle quali hanno dei valori default e altre no. Ovviamente le opzioni che non hanno un default vanno necessariamente fornite (come il percorso di un file in un comando che salva o apre un file). I valori di default, in genere sono abbastanza sensati e vanno cambiati solo se necessario. Curiosamente, i nomi delle opzioni non sono sempre necessari (anche se è meglio scriverli, per rendere più facilmente interpretabile il codice). Questi comandi sono equivalenti:
write_tsv(Arthritis, file = "Arthritis.tsv", append = F)
write_tsv(Arthritis, "Arthritis.tsv")
write_tsv(Arthritis, na = "NA", file = "Arthritis.tsv")Non è necessario dare un nome ad un’opzione se compare esattamente nella sequenza prevista dal comando. E’ anche possibile indicare le opzioni in disordine, ma in questo caso è necessario usare il nome.
5.9 Appendice 2. Se sei arrivat* fin qui…
La ragione principale per imparare a usare R dovrebbe essere quella di risolvere alcuni problemi in maniera efficiente ed automatizzata. Il numero di casi speciali che ti potresti trovare ad affrontare è praticamente infinito. E’ abbastanza comune trovarsi nella situazione di dover importare molti file con la stessa struttura in un unico data frame. L’operazione può essere condotta in maniera interattiva, ma con un elevato rischio di errore e poca riproducibilità. Cerca di capire cosa fa questo piccolo script (usando l’aiuto quando incontri un comando che non conosci) e poi prova ad eseguirlo (ometto il prompt per facilitarti le cose)123:
# preparazione, divido Arthritis in k=5 pezzi
j = 5
if(j>nrow(Arthritis)) cat("\nATTENZIONE: scegli un valore più piccolo per k\n")
# crea opzionalmente una directory per contenere i file
if(!("kArthritis" %in% list.files(file.path(".")))) dir.create(file.path(".","kArthritis"))
# crea un indice per le righe dei pezzi da salvare
indice <- sort(gl(j, 1, nrow(Arthritis)))
# creo una copia
Arthritis_3 <- Arthritis
Arthritis_3$indice <- indice
# uso un loop (più avanti vedremo come questo può essere sostituito da un functional) per
# salvare pezzi separati
for (i in seq_along(1:j)){
minidf <- dplyr::filter(Arthritis_3, indice == i)
nome_file <- str_c("Arthritis_",i,".tsv", sep ="")
write_tsv(dplyr::select(minidf, -indice), file = file.path(".", "kArthritis", nome_file))
}
# ora uso un loop per rimettere insieme i pezzi, usando i nomi dei file come indice
# quanti file di tipo .tsv ci sono?
nomi_file <- list.files(file.path(".", "kArthritis"))
quali_sono_tsv <- str_detect(nomi_file, "\\.tsv")
nomi_file<-nomi_file[quali_sono_tsv]
# creo una list vuota della lunghezza giusta
lista_file <- vector("list", length = length(nomi_file))
# questo loop legge i nomi dei file negli slot della lista
for (i in seq_along(nomi_file)){
lista_file[[i]] <- read_tsv(file.path(".","kArthritis", nomi_file[i]))
names(lista_file)[i]<-nomi_file[i]
}
# trasforma la lista in un data frame (e se si potesse fare con un loop?)
# modo n. 1, bruttino
Arthritis_frankenstein_brutto <- do.call(rbind, lista_file)
# modo n. 2 con map_dfr(), un functional, cioé un comando che applica una funzione a una lista
# e restituisce un data frame
Arthritis_frankenstein <- map_dfr(lista_file, bind_rows) # senza conservare i nomi dei file
Arthritis_frankenstein_nomi <- map_dfr(lista_file, bind_rows, .id = "nomi_file") # con i nomi dei file
# e, per farla ancora più semplice
percorsi <- file.path(".","kArthritis", nomi_file)
Arthritis_frankenstein_nomi_2 <- map_dfr(percorsi, read_tsv, .id = "nomi_file")Perché darsi tutta questa pena a scrivere tante righe di codice per fare una cosa che si poteva fare in modo interattivo? Intanto, si può scrivere codice in modo succinto. E poi, perché magari i file potevano essere 1000…, e anche perché magari ti potresti trovare a fare la stessa cosa cento volte e non la vuoi fare ogni volta in un modo diverso (in fondo, sei pigr*). E anche e soprattutto perché è più stimolante così. Naturalmente, nel mondo reale potevano andare male un sacco di cose e un programmatore prudente dovrebbe tenerne conto creando delle “trappole per gli errori”: se qualcosa non va come previsto, è bene che una funzione o una serie di comandi restituiscano il risultato che rappresenta il miglior compromesso possibile, magari insieme ad un messaggio di errore o warning facile da capire.
5.10 Altre risorse.
Il materiale in questo capitolo è presentato in molte delle risorse citate nel Capitolo 4. Qui riporto solo le risorse nuove.
5.10.1 Risorse in inglese.
Documenti e pagine web.
Riporto qui alcuni libri e documenti disponibili online nei quali potrai trovare capitoli rilevanti per il contenuto di questo capitolo:
Libri: provate questi
questo capitolo riassume un po’ tutto quello che c’è da sapere sull’importazione (ordinata) di dati
come sempre R for data science fornisce esempi ed esercizi interessanti
documenti e siti web:
questa guida ufficiale del CRAN all’importazione di dati è completa e aggiornata
questo è un articolo interessante sul web scraping (come importare contenuti dal web) con R
questo è uno dei tanti articoli avanzati per gestire data set molto grandi (e che potrebbero eccedere la RAM) con R. Con poca fatica potete trovare molte risorse simili. Anche questo articolo fornisce alcune soluzioni interessanti. Pacchetti o funzioni più o meno specializzate, come
data.table::fread()o le funzioni dibigreadrpossono servire per affrontare problemi di importazione veramente grossi.
Questa è decisamente la scelta migliore per condividere file con altri.↩︎
è possibile cambiare questo default nelle preferenze di RStudio↩︎
ricordati di omettere il carattere per il prompt, >↩︎
Attenzione a creare progetti in cartelle di Google Drive se usate l’app di Google drive: in qualche caso ci sono problemi di sincronizzazione, specialmente nella gestione di alcuni tipi di documenti dinamici, come i notebook. Dropbox funziona molto meglio↩︎
dovrebbe essere semplice e intuitivo ma, se non riesci, guarda questo video↩︎
o incollare direttamente il percorso del file↩︎
personalmente io preferisco settare l’intero sistema con un locale con punto decimale come US o UK↩︎
in effetti un vettore carattere con i tipi delle diverse colonne↩︎
e magari fare qualche sforzo in più per capire come usare i percorsi dei file↩︎
una tabella scaricata da uno dei miei repository su GitHub↩︎
se usi solo il nome del file con l’estensione appropriata il file verrà cercato nella working directory, vedi nei paragrafi precedenti. Puoi anche specificare un percorso diverso, vedi dopo.
fileè, in effetti, un vettore carattere di lunghezza 1 e puoi crearlo con una funzione!↩︎se non la attribuisci ad un oggetto la visualizzi soltanto in console!↩︎
e quindi da quasi tutti gli altri software per fogli di calcolo, che in genere hanno funzioni di importazione per questo formato↩︎
come al solito, sia che tu li esegua dalla console che che li esegua come uno script ricordati di cancellare
>: il prompt appare automaticamente nella console e non è necessario negli script↩︎in data frame↩︎
quindi con il nome fra virgolette; questo può essere comodo in alcune circostanze, quando i nomi vengono ricavati da un comando che produce, come risultato, una stringa di caratteri↩︎
come avrai potuto notare, i nomi delle funzioni del tidyverse sono un pochino più coerenti e facili da ricordare di quelli di R base: in
readr, tutte le funzioni sono write_xxx e read_xxx: come conseguenza quando inizi a digitare in RStudio i suggerimenti che ottieni sono un pochino più appropriati↩︎facendo doppio click sul nome dell’oggetto corrispondente nel pannello dell’ambiente o usando il comando
View()↩︎pillarviene caricato come dependency quando carichi il pacchettotidyverse↩︎se non usi menu interattivi nel report non è possibile aprire manualmente file o cambiare la cartella di lavoro↩︎
lo script assume che i file di testo che vuoi aprire abbiano tutti la stessa struttura in colonne↩︎